Classification of Small Bowel Lesions in Video Capsule Endoscopy Images Using Enhanced Deep Learning Model
DOI:
https://doi.org/10.17488/RMIB.46.SI-TAIH.1526Keywords:
convolutional neural network, deep learning, small bowel lesions, synthetic oversampling, video capsule endoscopyAbstract
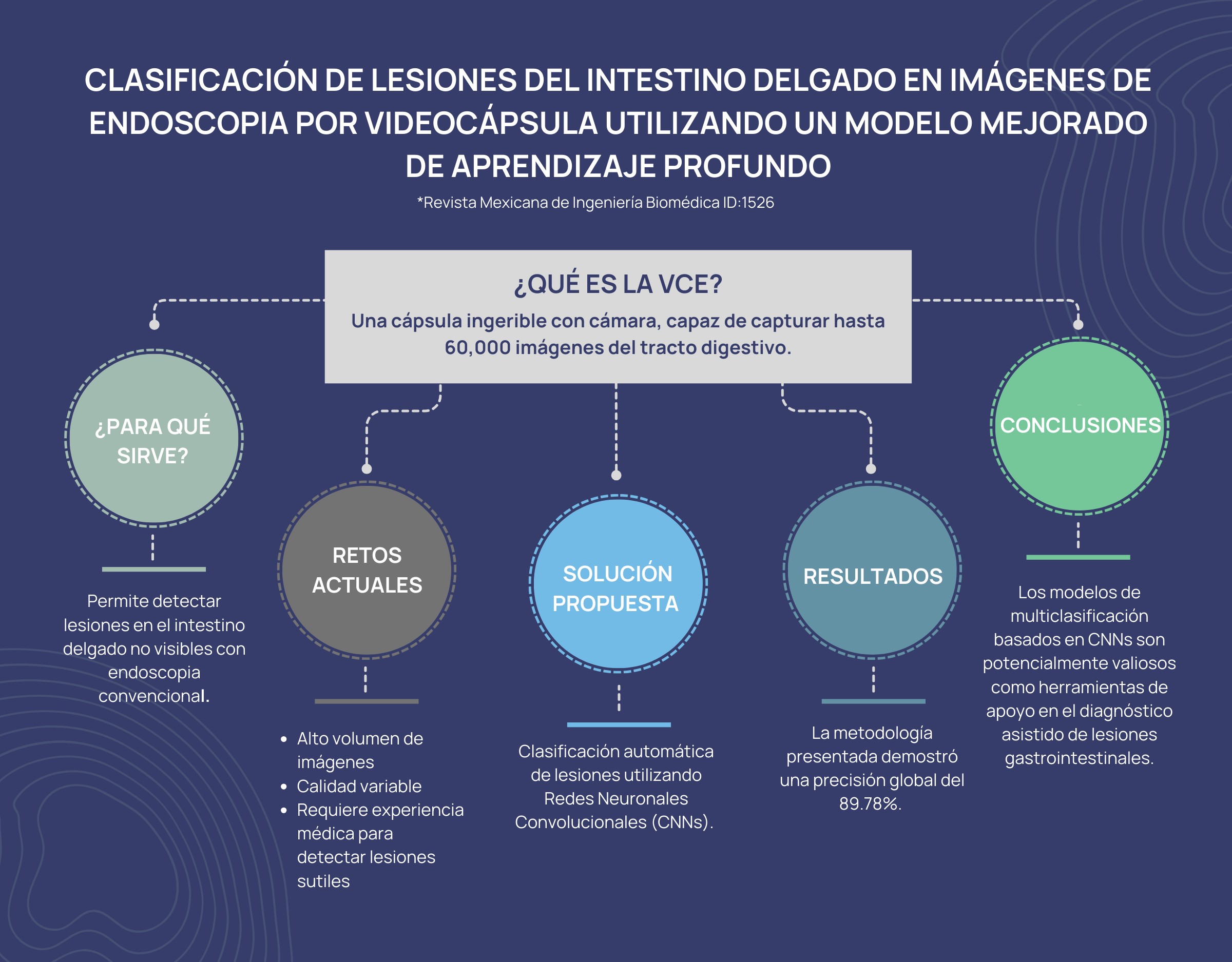
Video capsule endoscopy (VCE) is a noninvasive procedure for diagnosing small bowel (SB) lesions. During VCE procedures, the miniature camera captures thousands of images, requiring considerable time and effort from healthcare professionals to examine each image for abnormalities. This study aims to develop and evaluate a modified DenseNet-201 convolutional neural network (CNN) model to assist in the automatic classification of SB lesions in VCE images. A representative dataset of 5,899 images was created by merging two state-of-the-art datasets, including six types of lesions and one class without lesions. A synthetic oversampling approach generated 1,273 synthetic images for the underrepresented class. Synthetic image quality was evaluated using texture metrics. The experiment was conducted on a set of 7,172 images using data partitions of 70 %, 15 %, and 15 % for training, validation, and testing, respectively, with a Monte Carlo cross-validation to verify the consistency of the experimental results. The model achieved an accuracy of 89.78 %, a precision of 89.66 %, a sensitivity of 89.69 %, a specificity of 89.72 %, and an F1 score of 89.67 % in the test set. The modified DenseNet-201 CNN model could be a valuable tool for diagnosing SB conditions and improving patient outcomes.
Downloads
References
M. F. Lynch Mejia, “La cápsula endoscópica como estudio diagnóstico en gastroenterología,” Rev. Med. Sinerg., vol. 4, no. 4, pp. 18-25, 2019, doi: https://doi.org/10.31434/rms.v4i4.179
J. A. Urrego, W. O. Regino, and M. G. Zuleta, “Is the videocapsule endoscopy the best option for diagnosis of possible bleeding from the small intestine?,” Rev. Colomb. Gastroenterol., vol. 35, no. 2, pp. 196–206, 2020, doi: https://doi.org/10.22516/25007440.262
J. Melson et al., “Video capsule endoscopy,” Gastrointest. Endosc., vol. 93, no. 4, pp. 784–796, 2021, doi: https://doi.org/10.1016/J.GIE.2020.12.00
D. Marin-Santos, et. al., “Automatic detection of Crohn disease in wireless capsule endoscopic images using a deep convolutional neural network,” Appl. Intell., vol. 53, no. 10, pp. 12632–12646, 2022, doi: https://doi.org/10.1007/s10489-022-04146-3
E. Klang et al., “Automated detection of Crohn’s Disease intestinal strictures on capsule endoscopy images using deep neural networks,” J. Crohns Colitis, vol. 15, no. 5, pp. 749–756, 2021, art. no. 103638, doi: https://doi.org/10.1093/ecco-jcc/jjaa234
Ş. Öztürk and U. Özkaya, “Residual LSTM layered CNN for classification of gastrointestinal tract diseases,” J. Biomed. Inform., vol. 113, 2021, art. no. 103638, doi: https://doi.org/10.1016/j.jbi.2020.103638
T. Agrawal, R. Gupta, S. Sahu, and C. Espy-Wilson, “SCL-UMD at the medico task-mediaeval 2017: Transfer learning based classification of medical images,” in CEUR Workshop Proc., vol. 1984, Dublin, Ireland, 2017, pp. 3–5. [Online]. Available: https://ceur-ws.org/Vol-1984/Mediaeval_2017_paper_21.pdf
Y. Chang, Z. Huang, W. Chen and Q. Shen, “Gastrointestinal tract diseases detection with deep attention neural network,” in Proc. of the 27th ACM International Conference on Multimedia, New York, NY, US, 2019, pp. 2568–2572, doi: https://doi.org/10.1145/3343031.3356061
T. H. Hoang, et al., “An application of residual network and faster - RCNN for medico: Multimedia task at MediaEval 2018,” in CEUR Workshop Proc, vol. 2283, Sophia Antipolis, France, 2018, pp. 3–5. [Online]. Available: https://ceur-ws.org/Vol-2283/MediaEval_18_paper_11.pdf
Z. M. Lonseko et al., “Gastrointestinal Disease Classification in Endoscopic Images Using Attention-Guided Convolutional Neural Networks,” Appl. Sci., vol. 11, no. 23, 2021, art. no. 11136, doi: https://doi.org/10.3390/APP112311136
T. Rahim, M. A. Usman, and S. Y. Shin, “A survey on contemporary computer-aided tumor, polyp, and ulcer detection methods in wireless capsule endoscopy imaging,” Comput. Med. Imag. Graph., vol. 85, 2020, art. no. 101767, doi: https://doi.org/10.1016/J.COMPMEDIMAG.2020.101767
A. Caroppo, A. Leone, and P. Siciliano, “Deep transfer learning approaches for bleeding detection in endoscopy images,” Comput. Med. Imag.Graph., vol. 88, 2021, art. no. 101852, doi: https://doi.org/10.1016/J.COMPMEDIMAG.2020.101852
X. Wang et al., “Celiac disease diagnosis from videocapsule endoscopy images with residual learning and deep feature extraction,” Comput. Methods Programs Biomed., vol. 187, 2020, art. no. 105236, doi: https://doi.org/10.1016/J.CMPB.2019.105236
Y. Shin, et al., “Automatic Colon Polyp Detection Using Region Based Deep CNN and Post Learning Approaches,” IEEE Access, vol. 6, pp. 40950–40962, 2018, doi: https://doi.org/10.1109/ACCESS.2018.2856402
A. Nogueira-Rodríguez et al., “Deep Neural Networks approaches for detecting and classifying colorectal polyps,” Neurocomput., vol. 423, pp. 721–734, 2021, doi: https://doi.org/10.1016/J.NEUCOM.2020.02.123
M. Souaidi and M. El Ansari, “Multi-scale hybrid network for polyp detection in wireless capsule endoscopy and colonoscopy images,” Diagnostics, vol. 12, no. 8, 2022, art. no. 2030, doi: https://doi.org/10.3390/DIAGNOSTICS12082030
P. M. Vieira, et al., “Multi-pathology detection and lesion localization in WCE videos by using the instance segmentation approach,” Artif. Intell. Med., vol. 119, 2021, art. no. 102141, doi: https://doi.org/10.1016/j.artmed.2021.102141
F. Fonseca, B. Nunes, M. Salgado, and A. Cunha, “Abnormality classification in small datasets of capsule endoscopy images,” Procedia Comput. Sci., vol. 196, pp. 469–476, 2022, doi: https://doi.org/10.1016/J.PROCS.2021.12.038
S. Mahmood et al., “A robust deep model for classification of peptic ulcer and other digestive tract disorders using endoscopic images,” Biomedicines, vol. 10, no. 9, 2022, art. no. 2195, doi: https://doi.org/10.3390/BIOMEDICINES10092195
S. Jain et al., “A deep CNN model for anomaly detection and localization in wireless capsule endoscopy images,” Comput. Biol. Med., vol. 137, 2021, art. no. 104789, doi: https://doi.org/10.1016/j.compbiomed.2021.104789
Y. Ku, H. Ding, and G. Wang, “Efficient Synchronous real-time CADe for multicategory lesions in gastroscopy by using multiclass detection model,” Biomed. Res. Int., vol. 2022, 2022, art. no. 504149, doi: https://doi.org/10.1155/2022/8504149
P. H. Smedsrud et al., “Kvasir-Capsule, a video capsule endoscopy dataset,” Sci. Data, vol. 8, no. 1, 2021, art. no. 142, doi: https://doi.org/10.1038/s41597-021-00920-z
R. Vallée, et al., “CrohnIPI: An endoscopic image database for the evaluation of automatic Crohn’s disease lesions recognition algorithms,” in Proc. SPIE 11317, Medical Imaging 2020: Biomedical Applications in Molecular, Structural, and Functional Imaging, Houston, TX, US., 2020, pp. 440–446, doi: https://doi.org/10.1117/12.2543584
A. C. Bovik, “Chapter 3-Basic Gray Level Image Processing,” in The Essential Guide to Image Processing, 2nd. Ed., 2009, pp. 43–68, doi: https://doi.org/10.1016/B978-0-12-374457-9.00003-2
E. Mocsari and S. S. Stone, “Densely Connected Convolutional Networks,” 2017, arXiv:1608.06993, doi: https://doi.org/10.48550/arXiv.1608.06993
H. He, Y. Bai, E. A. Garcia, and S. Li, “ADASYN: Adaptive synthetic sampling approach for imbalanced learning,” in Proc. of the International Joint Conference on Neural Networks (IEEE World Congress on Computational Intelligence), Hong Kong, China, 2008, pp. 1322–1328, doi: https://doi.org/10.1109/IJCNN.2008.4633969
E. O. Cuevas-Rodriguez et al., “Comparative study of convolutional neural network architectures for gastrointestinal lesions classification,” PeerJ, vol. 11, 2023, art. no. e14806, doi: https://doi.org/10.7717/PEERJ.14806
T. Chauhan, H. Palivela, and S. Tiwari, “Optimization and fine-tuning of DenseNet model for classification of COVID-19 cases in medical imaging,” Int. J. Inf. Manag. Data Insights, vol. 1, no. 2, 2021, art. no. 100020, doi: https://doi.org/10.1016/J.JJIMEI.2021.100020
O. Russakovsky et al., “ImageNet large scale visual recognition challenge,” Int. J. Comput. Vis., vol. 115, no. 3, pp. 211–252, 2015, doi: https://doi.org/10.1007/s11263-015-0816-y
A. de Maissin et al., “Multi-expert annotation of Crohn’s disease images of the small bowel for automatic detection using a convolutional recurrent attention neural network,” Endosc. Int. Open, vol. 9, no. 7, pp. E1136–E1144, 2021, doi: https://doi.org/10.1055/A-1468-3964
Downloads
Published
How to Cite
Issue
Section
License
Copyright (c) 2025 Revista Mexicana de Ingenieria Biomedica

This work is licensed under a Creative Commons Attribution-NonCommercial 4.0 International License.
Upon acceptance of an article in the RMIB, corresponding authors will be asked to fulfill and sign the copyright and the journal publishing agreement, which will allow the RMIB authorization to publish this document in any media without limitations and without any cost. Authors may reuse parts of the paper in other documents and reproduce part or all of it for their personal use as long as a bibliographic reference is made to the RMIB. However written permission of the Publisher is required for resale or distribution outside the corresponding author institution and for all other derivative works, including compilations and translations.







